文献阅读-20230704-多种植物中根系发育的保守基因表达程序
1. 背景
文章标题:Conserved Gene Expression Programs in Developing Roots from Diverse Plants
发表时间:20150811
发表期刊:The Plant Cell
一作及通讯:Ling Huang(上海交大本硕微生物,之后去联合利华研发洁面乳一年,然后去密歇根大学读博士,中途去孟山都公司做了rnaseq的pipeline,然后去Salk干了5年,现在在Neomorph公司,感谢Linkedin平台🙏), John Schiefelbein(现于密歇根大学,实验室网站https://sites.lsa.umich.edu/schiefelbein-lab/)
原文链接:https://doi.org/10.1105/tpc.15.00328
2. 摘要
The molecular basis for the origin and diversification of morphological adaptations is a central issue in evolutionary developmental biology. Here, we defined temporal transcript accumulation in developing roots from seven vascular plants, permitting a genome-wide comparative analysis of the molecular programs used by a single organ across diverse species. The resulting gene expression maps uncover significant similarity in the genes employed in roots and their developmental expression profiles. The detailed analysis of a subset of 133 genes known to be associated with root development in Arabidopsis thaliana indicates that most of these are used in all plant species. Strikingly, this was also true for root development in a lycophyte (Selaginella moellendorffii), which forms morphologically different roots and is thought to have evolved roots independently. Thus, despite vast differences in size and anatomy of roots from diverse plants, the basic molecular mechanisms employed during root formation appear to be conserved. This suggests that roots evolved in the two major vascular plant lineages either by parallel recruitment of largely the same developmental program or by elaboration of an existing root program in the common ancestor of vascular plants.
形态适应性起源和多样化的分子基础是进化发育生物学的核心问题。在这里,我们定义了七种维管植物发育根中的时间转录累积,允许进行跨物种的全基因组比较分析,研究单一器官在不同物种中使用的分子程序。由此产生的基因表达图揭示了根和其发育表达谱中所使用的基因的显著相似性。对133个已知与拟南芥根发育相关的基因的详细分析表明,这些基因大多数在所有植物物种中都被使用。令人惊讶的是,尽管石松类植物(Selaginella moellendorffii)形成了形态不同的根,并被认为独立进化了根,但这也适用于蕨类植物中的根发育。因此,尽管不同植物的根的大小和解剖结构差异巨大,但在根形成期间使用的基本分子机制似乎是保守的。这表明,两个主要维管植物系的根要么通过并行招募大部分相同的发育程序,要么通过在维管植物的共同祖先中发展现有的根程序来进化。
3. Figure
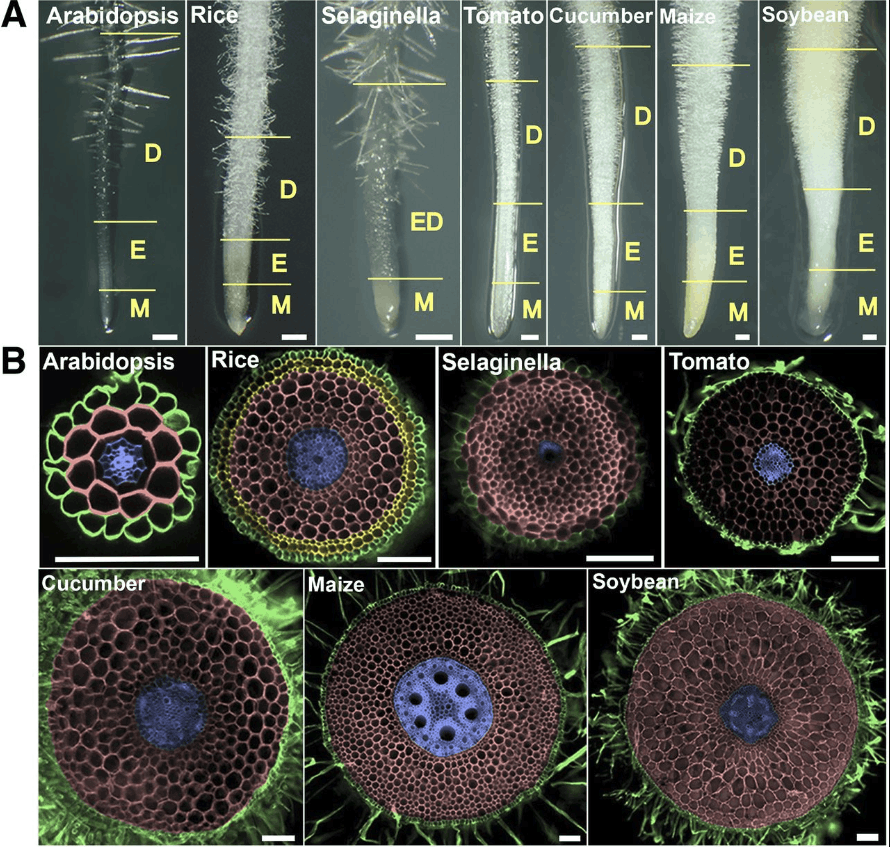
- fig.1 拟南芥、水稻、江南卷柏、番茄、黄瓜、玉米、大豆根系图与横切后显微镜图
- 其中根系基本都分为分生区M,伸长区E,分化区D,唯独在江南卷柏中伸长区和分化区合并为一个区ED。
- fig.2 拟南芥RNA-seq结果与已发表的微阵列数据的皮尔逊相关性。
- fig.3 六种被子植物根的三个发育区域的基因表达偏好性。
- A,在不同物种的根发育区中表达基因的比例(平均FPKM>=0.5并且至少两个重复有表达时,视为该基因表达)。
- B,每个物种单独进行PCA分析后,所有物种的PCA结果绘制到一张图中。
从这里开始图就很难看懂了
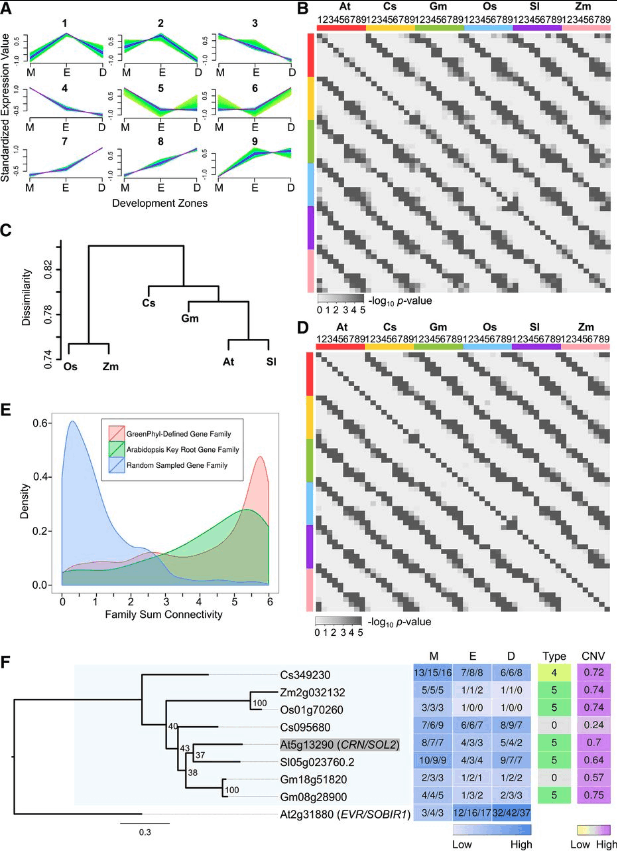
- fig.4 被子植物间根基因表达的比较。
- A,使用fuzzy C-Means聚类,按基因在M、E、D三个区域的表达情况分为了9类(就是mfuzz包,只对在任意两个区之间foldchange大于等于2的基因分类)。
- B,在六个物种中都按A图的方法分了9类基因,然后统计不同物种不同类两两之间是否属于同一个家族。
- C,某个特定家族的重叠表达类型的平均比率与已知的物种进化树相同。
- D,与B类似,只是把基因家族gene families换成了超基因家族supergene families,举个例子,一个家族内拟南芥有10个基因,把这10个基因的表达量加起来作为代表拟南芥在这个家族的supergene,然后再按图A计算表达类型。
- E,很多家族总连通性的密度图,每个物种中计算该物种在该家族的supergene,与该物种在这个家族的所有基因的CNV,每个家族的连通性为6个物种相加,所以横坐标最大为6。红/绿/蓝分别代表了,6161个来自六个被子植物由greenphy定义的根表达超基因家族,73个含有拟南芥已知的关键根发育基因由greenphy定义的家族,以及1000个随机分配家族成员的基因家族。
- F,用拟南芥已知的关键根发育基因构建了71个进化树,这是其中一个,物种为被子植物,at拟南芥,cs黄瓜,gm大豆,os水稻,sl番茄,zm玉米。MED是FPKM表达热图,每个方块里面的数字代表三次重复;Type是这个基因的表达类型,对应图A,CNV,connectivity values(CNV was calculated as the average pairwise Pearson's correlation coefficient of a given gene's expression profile to all the genes’ profiles within a given family)。
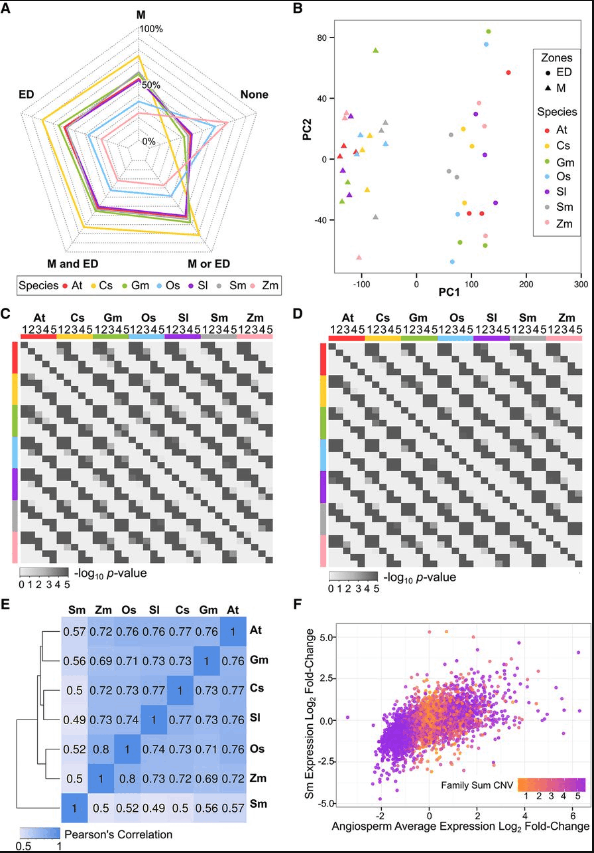
- fig.5
- A和B,在fig.3 A&B的基础上添加了sm江南卷柏的表达信息。
- C和D,类似于fig.4 C&D,添加了sm江南卷柏的表达信息。
- E,5027个GreenPhyl定义的基因家族中(5027的来源,在GreenPhyl定义的基因家族中,七个物种都各自至少有一个在根中表达的基因的基因家族有5027个),7个物种两两之间的相关性矩阵,通过(log2 EDZ/MZ FC)计算。
- F,散点图表示了5027个GreenPhyl定义的基因家族中,sm(log2 EDZ/MZ FC)和其他六个物种的(log2 EDZ/MZ FC)平均值的情况,颜色代表家族总的CNV,理论上最大值为7吧,这张图表现了两个值之间的强相关性(F-statistic P < 0.001)。
-
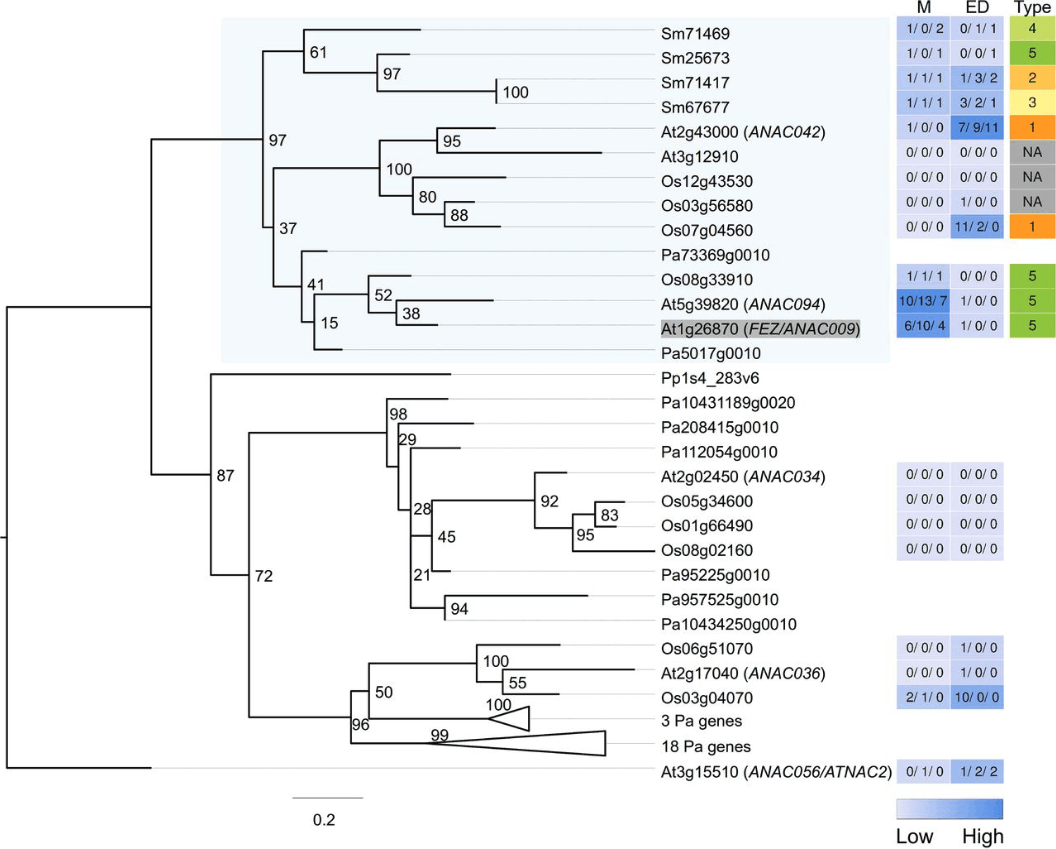
fig.6(文章好像完全没有提到fig6?感觉只放fig.7就可以了啊) 类似于fig.4 F,用拟南芥已知的关键根发育基因构建了71个进化树,这是其中关于AtGNOM的一个,物种在fig.4 F的基础上添加了sm江南卷柏,pa挪威云杉,pp小立碗藓
-
fig.7 与fig.6一样,是71个进化树之一,是关于AtFEZ(促进根冠干细胞活性)的一个。
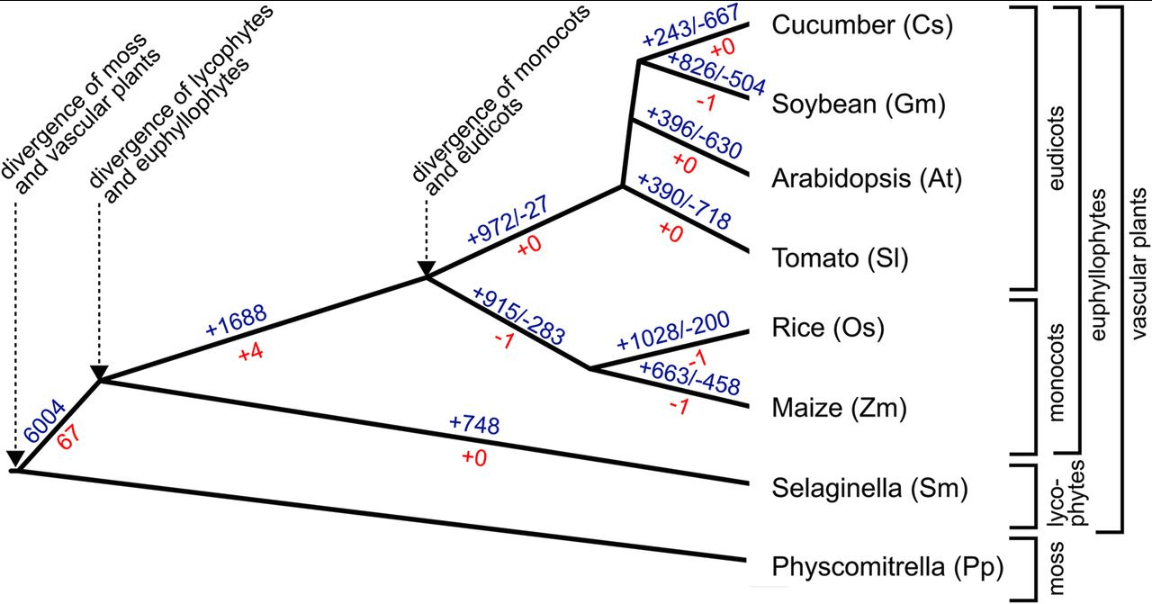
- fig.8 在根表达的基因家族的进化历史,蓝色数字代表GreenPhyl定义的根表达基因家族数量,红色代表拟南芥133个根发育相关基因所在的71个基因家族。蓝色红色的正负号数字代表各自的gain或者loss。
4. 后记
- 测了几个维管植物(拟南芥、水稻、江南卷柏、番茄、黄瓜、玉米、大豆)的分生区,伸长区,分化区的RNA-Seq
- 经过一通计算说明了石松植物和真叶植物的具有保守的根发育调控程序