FastQ Screen检测测序数据污染情况
1.前言
检测测序数据中有无污染
2.安装及测试
https://www.bioinformatics.babraham.ac.uk/projects/fastq_screen/
https://github.com/StevenWingett/FastQ-Screen
mamba activate fastqscreen
mamba install -y bowtie2
# 安装软件
cd ~/software
wget https://github.com/StevenWingett/FastQ-Screen/archive/refs/tags/v0.15.3.tar.gz
tar -zxvf v0.15.3.tar.gz
# perl -MCPAN -e "install GD"
# 下载测试数据
mkdir test && cd test
wget https://www.bioinformatics.babraham.ac.uk/projects/fastq_screen/fastq_screen_test_dataset.tar.gz
tar xvzf fastq_screen_test_dataset.tar.gz
# 下载常用基因组索引
fastq_screen --get_genomes # 会下载到当前目录的FastQ_Screen_Genomes下,我是~/software/FastQ-Screen-0.15.3/test/FastQ_Screen_Genomes
# 另外上面这个目录下面还会有一个fastq_screen.conf文件,直接写好了基因组索引地址,用起来很方便
# 对这个conf文件简单修改一下线程40和bowtie2地址即可
# test run
~/software/FastQ-Screen-0.15.3/fastq_screen -c FastQ_Screen_Genomes/fastq_screen.conf fastq_screen_test_dataset/fqs_test_dataset.fastq.gz
# 运行完成后会在当前目录下生成两个文件
# fqs_test_dataset_screen.html
# fqs_test_dataset_screen.txt
3.运行自己的数据
cd /mnt/caigui/77_QHT_clear_assemble/5_fastqscreen
~/software/FastQ-Screen-0.15.3/fastq_screen \
-c ~/software/FastQ-Screen-0.15.3/test/FastQ_Screen_Genomes/fastq_screen.conf \
/mnt/caigui/74_QHT/2_kmer/2_default/03_fastp/E100074680_L01_1_fastp_R1.fastq.gz \
/mnt/caigui/74_QHT/2_kmer/2_default/03_fastp/E100074680_L01_1_fastp_R2.fastq.gz
remind.sh fastqscreen_end
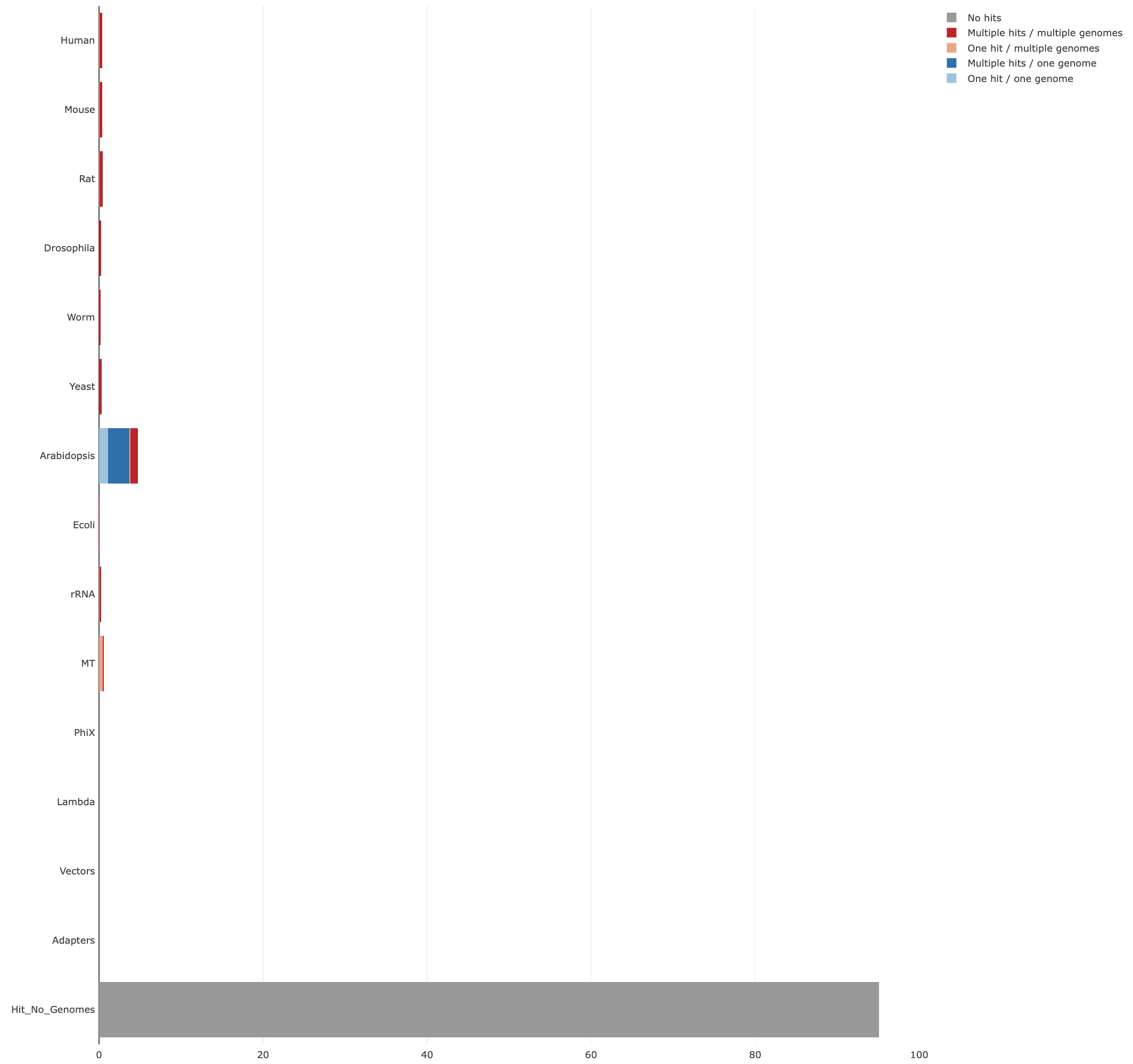
# 每个fastq.gz都会生成一个单独的html和txt结果文件,下面放一个举例
# 默认是选10万条reads去做比对,所以速度挺快的。当然比对数量也可以在参数里调整。
# 从二代结果来看,我这套数据污染程度不高,有一些线粒体污染